T: Detección de Chlamydia pneumoniae en tejido
aórtico humano: amplificación del gen kdtA e hibridación in vitro.
O: Diagnóstico.
Investigar la presencia de C. pneumoniae en muestras de tejido
aórtico de catorce pacientes sometidos a cirugía de reemplazo aórtico,
utilizando la amplificación del genkdtA por PCR acoplada a un
ensayo de hibridación in vitro.
M: Tejido
aórtico obtenido de catorce pacientes con diagnóstico de enfermedad de aorta
ascendente en un frasco estéril que contenía una solución de etanol al 70%. (Las
muestras fueron almacenadas a 4°C hasta el momento de la extracción de ADN).
AN: ADN bacteriano.
La extracción de ADN a partir de tejido de aorta se estandarizó utilizando 4
muestras que fueron sometidas a procedimientos de extracción con dos estuches
comerciales (QIAamp DNA Mini Kit de QIAGEN® y AquaPure Genomic DNA kit de
BIORAD ®) de acuerdo con las recomendaciones de los fabricantes. El ADN de
las 10 arterias restantes se extrajo utilizando el estuche AquaPure Genomic DNA
de BIO-RAD®.
G: Gen kdtA.
PCR: PCR Estándar.
Se empleó el estuche/kit: Light DiagnosticsTM Chlamydia
pneumoniae Oligodetect de Chemicon®, de acuerdo con las
recomendaciones del fabricante.
Perfil térmico:
40 ciclos de denaturación, anillaje y elongación a 94°C (1 min), Denaturación
inicial de 1 minuto a 94°C; 58°C (30 seg) y 72°C (1 min), respectivamente, y 8
minutos a 72°C para la elongación final.
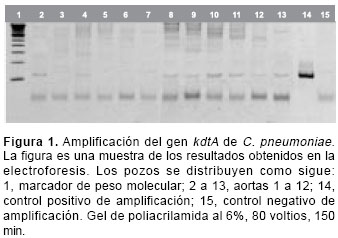
V: Por
electroforesis en geles de poliacrilamida al 6% en corrido de 150 minutos a 80
voltios.
Control Negativo de Amplificación: 1ml de agua ultrapura en lugar de ADN.
Control Positivo:
Se utilizó el que provee el estuche: DiagnosticsTM Chlamydia
pneumoniae Oligodetect de Chemicon®.
Se
señala que la técnica es muy sensible y específica, pero no se dan valores
analíticos.
“…la
técnica es altamente específica para la detección de C. pneumoniae,
ya que se ha reportado (19) que la secuencia de aminoácidos deducida de la
secuenciación de nucleótidos del genkdtA de C. pneumoniae amplificado
por PCR en nuestro estudio tiene 69% de similitud y 43% de identidad con la
secuencia correspondiente en C. trachomatis y C.
psittaci (las otras dos especies que afectan al ser humano), lo cual
hace de esta secuencia un candidato muy apropiado para la detección de C.
pneumoniae debido a que no es de esperarse que existan reacciones
cruzadas con las otras dos especies mencionadas.”
Se
evitó la presencia de falsos positivos y negativos
pues se trabajo juntamente con una técnica de hibridación in vitro.
ARTÍCULO: Detección de Chlamydia pneumoniae en tejido aórtico humano.